dendrogram#
- scipy.cluster.hierarchy.dendrogram(Z, p=30, truncate_mode=None, color_threshold=None, get_leaves=True, orientation='top', labels=None, count_sort=False, distance_sort=False, show_leaf_counts=True, no_plot=False, no_labels=False, leaf_font_size=None, leaf_rotation=None, leaf_label_func=None, show_contracted=False, link_color_func=None, ax=None, above_threshold_color='C0')[Quelle]#
Zeichnet das hierarchische Clustering als Dendrogramm.
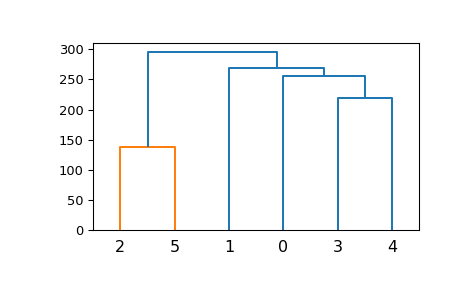
Das Dendrogramm veranschaulicht, wie jeder Cluster aufgebaut ist, indem es eine U-förmige Verbindung zwischen einem nicht-einzelnen Cluster und seinen Kindern zeichnet. Die Spitze des U-Links zeigt die Cluster-Zusammenführung. Die beiden Schenkel des U-Links geben an, welche Cluster zusammengeführt wurden. Die Länge der beiden Schenkel des U-Links repräsentiert den Abstand zwischen den Kind-Clustern. Es ist auch der kophänetische Abstand zwischen den ursprünglichen Beobachtungen in den beiden Kind-Clustern.
- Parameter:
- Zndarray
Die Linkage-Matrix, die das hierarchische Clustering kodiert, das als Dendrogramm gerendert werden soll. Weitere Informationen zum Format von
Zfinden Sie in der Funktionlinkage.- pint, optional
Der Parameter
pfürtruncate_mode.- truncate_modestr, optional
Das Dendrogramm kann schwer zu lesen sein, wenn die ursprüngliche Beobachtungsmatrix, aus der die Verknüpfung abgeleitet ist, groß ist. Die Trunkierung wird verwendet, um das Dendrogramm zu kondensieren. Es gibt mehrere Modi
NoneKeine Trunkierung wird durchgeführt (Standard). Hinweis:
'none'ist ein Alias fürNone, der aus Kompatibilitätsgründen beibehalten wird.'lastp'Die letzten
pnicht-einzelnen Cluster, die in der Verknüpfung gebildet wurden, sind die einzigen nicht-Blattknoten in der Verknüpfung; sie entsprechen den ZeilenZ[n-p-2:end]inZ. Alle anderen nicht-einzelnen Cluster werden in Blattknoten komprimiert.'level'Nicht mehr als
pEbenen des Dendrogrammbaums werden angezeigt. Eine „Ebene“ umfasst alle Knoten mitpZusammenführungen von der letzten Zusammenführung.Hinweis:
'mtica'ist ein Alias für'level', der aus Kompatibilitätsgründen beibehalten wird.
- color_thresholddouble, optional
Zur Kürze sei \(t\) der
color_threshold. Färbt alle Nachfolgelinks unterhalb eines Clusterknotens \(k\) gleich, wenn \(k\) der erste Knoten unterhalb des Schnittschwellenwerts \(t\) ist. Alle Links, die Knoten mit Abständen verbinden, die größer oder gleich dem Schwellenwert sind, werden mit der Standardfarbe von Matplotlib'C0'gefärbt. Wenn \(t\) kleiner oder gleich Null ist, werden alle Knoten'C0'gefärbt. Wenncolor_thresholdNone oder ‘default’ ist, was dem MATLAB(TM)-Verhalten entspricht, wird der Schwellenwert auf0.7*max(Z[:,2])gesetzt.- get_leavesbool, optional
Enthält eine Liste
R['leaves']=Him Ergebnis-Dictionary. Für jedes \(i\) giltH[i] == j, wobei der Clusterknotenjan der Positioniin der Links-nach-rechts-Durchquerung der Blätter erscheint, wobei \(j < 2n-1\) und \(i < n\).- orientationstr, optional
Die Richtung, in der das Dendrogramm gezeichnet wird, kann einer der folgenden Zeichenketten entsprechen
'top'Zeichnet die Wurzel oben und die Nachfolgelinks nach unten. (Standard).
'bottom'Zeichnet die Wurzel unten und die Nachfolgelinks nach oben.
'left'Zeichnet die Wurzel links und die Nachfolgelinks nach rechts.
'right'Zeichnet die Wurzel rechts und die Nachfolgelinks nach links.
- labelsndarray, optional
Standardmäßig ist
labelsNone, sodass der Index der ursprünglichen Beobachtung zur Beschriftung der Blattknoten verwendet wird. Andernfalls handelt es sich um eine \(n\)-große Sequenz, mitn == Z.shape[0] + 1. Der Wertlabels[i]ist der Text, der unter dem \(i\)-ten Blattknoten platziert wird, nur wenn er einer ursprünglichen Beobachtung und keinem nicht-einzelnen Cluster entspricht.- count_sortstr or bool, optional
Für jeden Knoten n wird die Reihenfolge (visuell von links nach rechts), in der die beiden Nachfolgelinks von n gezeichnet werden, durch diesen Parameter bestimmt, der einer der folgenden Werte sein kann
FalseEs wird nichts unternommen.
'ascending'oderTrueDas Kind mit der geringsten Anzahl von ursprünglichen Objekten in seinem Cluster wird zuerst gezeichnet.
'descending'Das Kind mit der größten Anzahl von ursprünglichen Objekten in seinem Cluster wird zuerst gezeichnet.
Hinweis:
distance_sortundcount_sortkönnen nicht beide True sein.- distance_sortstr or bool, optional
Für jeden Knoten n wird die Reihenfolge (visuell von links nach rechts), in der die beiden Nachfolgelinks von n gezeichnet werden, durch diesen Parameter bestimmt, der einer der folgenden Werte sein kann
FalseEs wird nichts unternommen.
'ascending'oderTrueDas Kind mit dem geringsten Abstand zwischen seinen direkten Nachfahren wird zuerst gezeichnet.
'descending'Das Kind mit dem größten Abstand zwischen seinen direkten Nachfahren wird zuerst gezeichnet.
Hinweis:
distance_sortundcount_sortkönnen nicht beide True sein.- show_leaf_countsbool, optional
Wenn True, werden Blattknoten, die \(k>1\) ursprüngliche Beobachtung repräsentieren, mit der Anzahl der enthaltenen Beobachtungen in Klammern beschriftet.
- no_plotbool, optional
Wenn True, wird das endgültige Rendering nicht durchgeführt. Dies ist nützlich, wenn nur die für das Rendering berechneten Datenstrukturen benötigt werden oder wenn Matplotlib nicht verfügbar ist.
- no_labelsbool, optional
Wenn True, erscheinen im Rendering des Dendrogramms keine Beschriftungen neben den Blattknoten.
- leaf_rotationdouble, optional
Gibt den Winkel (in Grad) für die Drehung der Blattbeschriftungen an. Wenn nicht angegeben, basiert die Drehung auf der Anzahl der Knoten im Dendrogramm (Standard ist 0).
- leaf_font_sizeint, optional
Gibt die Schriftgröße (in Punkten) der Blattbeschriftungen an. Wenn nicht angegeben, basiert die Größe auf der Anzahl der Knoten im Dendrogramm.
- leaf_label_funclambda oder function, optional
Wenn
leaf_label_funceine aufrufbare Funktion ist, wird sie für jedes Blatt mit dem Clusterindex \(k < 2n-1\) aufgerufen. Es wird erwartet, dass die Funktion eine Zeichenkette mit der Beschriftung für das Blatt zurückgibt.Indizes \(k < n\) entsprechen ursprünglichen Beobachtungen, während Indizes \(k \geq n\) nicht-einzelnen Clustern entsprechen.
Um beispielsweise Einzelne mit ihrer Knoten-ID und Nicht-Einzelne mit ihrer ID, Anzahl und ihrem Inkonsistenzkoeffizienten zu beschriften, tun Sie einfach
# First define the leaf label function. def llf(id): if id < n: return str(id) else: return '[%d %d %1.2f]' % (id, count, R[n-id,3]) # The text for the leaf nodes is going to be big so force # a rotation of 90 degrees. dendrogram(Z, leaf_label_func=llf, leaf_rotation=90) # leaf_label_func can also be used together with ``truncate_mode``, # in which case you will get your leaves labeled after truncation: dendrogram(Z, leaf_label_func=llf, leaf_rotation=90, truncate_mode='level', p=2)
- show_contractedbool, optional
Wenn True, werden die Höhen von nicht-einzelnen Knoten, die in einen Blattknoten komprimiert wurden, als Kreuze entlang der Verbindung dieses Blattknotens gezeichnet. Dies ist eigentlich nur nützlich, wenn eine Trunkierung verwendet wird (siehe Parameter
truncate_mode).- link_color_funccallable, optional
Wenn angegeben, wird die link_color_function für jede nicht-einzelne ID aufgerufen, die jedem zu malenden U-förmigen Link entspricht. Es wird erwartet, dass die Funktion die Farbe zurückgibt, mit der der Link gemalt werden soll, kodiert als Matplotlib-Farbzeichenkettencode. Zum Beispiel
dendrogram(Z, link_color_func=lambda k: colors[k])
färbt die direkten Links unter jedem nicht-trunkierten nicht-einzelnen Knoten
kunter Verwendung voncolors[k].- axmatplotlib Axes instance, optional
Wenn None und no_plot nicht True ist, wird das Dendrogramm auf den aktuellen Achsen gezeichnet. Andernfalls, wenn no_plot nicht True ist, wird das Dendrogramm auf der gegebenen
Axes-Instanz gezeichnet. Dies kann nützlich sein, wenn das Dendrogramm Teil einer komplexeren Abbildung ist.- above_threshold_colorstr, optional
Diese Matplotlib-Farbzeichenkette legt die Farbe der Links oberhalb des Farbschwellenwerts fest. Der Standardwert ist
'C0'.
- Rückgabe:
- Rdict
Ein Dictionary mit Datenstrukturen, die zur Darstellung des Dendrogramms berechnet wurden. Es hat die folgenden Schlüssel
'color_list'Eine Liste von Farbnamen. Das k-te Element repräsentiert die Farbe des k-ten Links.
'icoord'und'dcoord'Jedes davon ist eine Liste von Listen. Wenn
icoord = [I1, I2, ..., Ip], wobeiIk = [xk1, xk2, xk3, xk4]unddcoord = [D1, D2, ..., Dp], wobeiDk = [yk1, yk2, yk3, yk4], dann ist der k-te gezeichnete Link(xk1, yk1)-(xk2, yk2)-(xk3, yk3)-(xk4, yk4).'ivl'Eine Liste von Beschriftungen, die den Blattknoten entsprechen.
'leaves'Für jedes i gilt
H[i] == j, wobei der Clusterknotenjan der Positioniin der Links-nach-rechts-Durchquerung der Blätter erscheint, wobei \(j < 2n-1\) und \(i < n\). Wennjkleiner alsnist, entspricht deri-te Blattknoten einer ursprünglichen Beobachtung. Andernfalls entspricht er einem nicht-einzelnen Cluster.'leaves_color_list'Eine Liste von Farbnamen. Das k-te Element repräsentiert die Farbe des k-ten Blattes.
Siehe auch
Hinweise
Es wird erwartet, dass die Abstände in
Z[:,2]monoton sind, andernfalls treten Überkreuzungen im Dendrogramm auf.dendrogramhat experimentelle Unterstützung für Python Array API Standard-kompatible Backends zusätzlich zu NumPy. Bitte erwägen Sie, diese Funktionen zu testen, indem Sie eine UmgebungsvariableSCIPY_ARRAY_API=1setzen und CuPy-, PyTorch-, JAX- oder Dask-Arrays als Array-Argumente bereitstellen. Die folgenden Kombinationen von Backend und Gerät (oder anderer Fähigkeit) werden unterstützt.Bibliothek
CPU
GPU
NumPy
✅
n/a
CuPy
n/a
⛔
PyTorch
✅
⛔
JAX
⚠️ kein JIT
⛔
Dask
⚠️ berechnet Graph
n/a
Siehe Unterstützung für den Array API Standard für weitere Informationen.
Beispiele
>>> import numpy as np >>> from scipy.cluster import hierarchy >>> import matplotlib.pyplot as plt
Ein sehr einfaches Beispiel
>>> ytdist = np.array([662., 877., 255., 412., 996., 295., 468., 268., ... 400., 754., 564., 138., 219., 869., 669.]) >>> Z = hierarchy.linkage(ytdist, 'single') >>> plt.figure() >>> dn = hierarchy.dendrogram(Z)
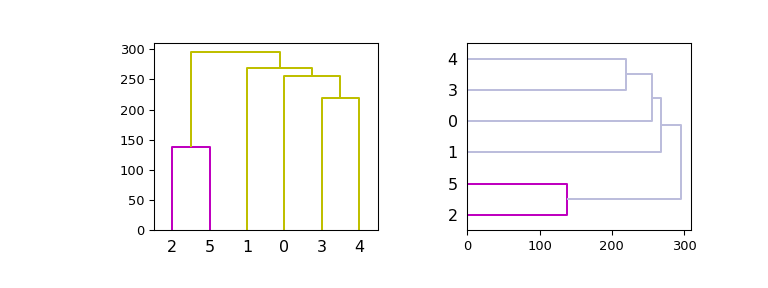
Nun, zeichnen Sie in gegebenen Achsen, verbessern Sie das Farbschema und verwenden Sie sowohl vertikale als auch horizontale Ausrichtungen
>>> hierarchy.set_link_color_palette(['m', 'c', 'y', 'k']) >>> fig, axes = plt.subplots(1, 2, figsize=(8, 3)) >>> dn1 = hierarchy.dendrogram(Z, ax=axes[0], above_threshold_color='y', ... orientation='top') >>> dn2 = hierarchy.dendrogram(Z, ax=axes[1], ... above_threshold_color='#bcbddc', ... orientation='right') >>> hierarchy.set_link_color_palette(None) # reset to default after use >>> plt.show()