yeojohnson_llf#
- scipy.stats.yeojohnson_llf(lmb, data)[Quelle]#
Die Yeo-Johnson Log-Likelihood-Funktion.
- Parameter:
- lmbSkalar
Parameter für die Yeo-Johnson-Transformation. Details siehe
yeojohnson.- dataarray_like
Daten, für die die Yeo-Johnson Log-Likelihood berechnet werden soll. Wenn data mehrdimensional ist, wird die Log-Likelihood entlang der ersten Achse berechnet.
- Rückgabe:
- llffloat
Yeo-Johnson Log-Likelihood von data gegeben lmb.
Siehe auch
Hinweise
Die Yeo-Johnson Log-Likelihood-Funktion \(l\) ist hier definiert als
\[l = -\frac{N}{2} \log(\hat{\sigma}^2) + (\lambda - 1) \sum_i^N \text{sign}(x_i) \log(|x_i| + 1)\]wobei \(N\) die Anzahl der Datenpunkte \(x`=``data`\) ist und \(\hat{\sigma}^2\) die geschätzte Varianz der Yeo-Johnson-transformierten Eingabedaten \(x\) ist. Dies entspricht der *Profil-Log-Likelihood* der ursprünglichen Daten \(x\), wobei einige konstante Terme weggelassen wurden.
Hinzugefügt in Version 1.2.0.
Beispiele
>>> import numpy as np >>> from scipy import stats >>> import matplotlib.pyplot as plt >>> from mpl_toolkits.axes_grid1.inset_locator import inset_axes
Einige Zufallsvariaten generieren und Yeo-Johnson Log-Likelihood-Werte für diese für einen Bereich von
lmbda-Werten berechnen.>>> x = stats.loggamma.rvs(5, loc=10, size=1000) >>> lmbdas = np.linspace(-2, 10) >>> llf = np.zeros(lmbdas.shape, dtype=float) >>> for ii, lmbda in enumerate(lmbdas): ... llf[ii] = stats.yeojohnson_llf(lmbda, x)
Auch den optimalen lmbda-Wert mit
yeojohnsonfinden.>>> x_most_normal, lmbda_optimal = stats.yeojohnson(x)
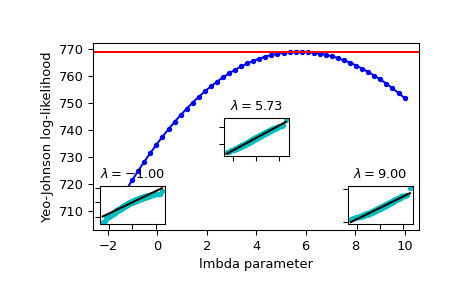
Die Log-Likelihood als Funktion von lmbda plotten. Die optimale lmbda als horizontale Linie hinzufügen, um zu überprüfen, ob dies wirklich das Optimum ist.
>>> fig = plt.figure() >>> ax = fig.add_subplot(111) >>> ax.plot(lmbdas, llf, 'b.-') >>> ax.axhline(stats.yeojohnson_llf(lmbda_optimal, x), color='r') >>> ax.set_xlabel('lmbda parameter') >>> ax.set_ylabel('Yeo-Johnson log-likelihood')
Nun einige Wahrscheinlichkeitsdiagramme hinzufügen, um zu zeigen, dass dort, wo die Log-Likelihood maximiert wird, die mit
yeojohnsontransformierten Daten am ehesten normal aussehen.>>> locs = [3, 10, 4] # 'lower left', 'center', 'lower right' >>> for lmbda, loc in zip([-1, lmbda_optimal, 9], locs): ... xt = stats.yeojohnson(x, lmbda=lmbda) ... (osm, osr), (slope, intercept, r_sq) = stats.probplot(xt) ... ax_inset = inset_axes(ax, width="20%", height="20%", loc=loc) ... ax_inset.plot(osm, osr, 'c.', osm, slope*osm + intercept, 'k-') ... ax_inset.set_xticklabels([]) ... ax_inset.set_yticklabels([]) ... ax_inset.set_title(r'$\lambda=%1.2f$' % lmbda)
>>> plt.show()