leaves_list#
- scipy.cluster.hierarchy.leaves_list(Z)[Quelle]#
Gibt eine Liste von Blattknoten-IDs zurück.
Die Ausgabe entspricht dem Index des Beobachtungsvektors, wie er im Baum von links nach rechts erscheint. Z ist eine Linkage-Matrix.
- Parameter:
- Zndarray
Die als Matrix kodierte hierarchische Gruppierung. Z ist eine Linkage-Matrix. Weitere Informationen finden Sie unter
linkage.
- Rückgabe:
- leaves_listndarray
Die Liste der Blattknoten-IDs.
Siehe auch
dendrogramfür Informationen über die Dendrogrammstruktur.
Hinweise
leaves_listhat experimentelle Unterstützung für Backends, die mit dem Python Array API Standard kompatibel sind, zusätzlich zu NumPy. Bitte erwägen Sie, diese Funktionen zu testen, indem Sie die UmgebungsvariableSCIPY_ARRAY_API=1setzen und CuPy-, PyTorch-, JAX- oder Dask-Arrays als Array-Argumente übergeben. Die folgenden Kombinationen aus Backend und Gerät (oder anderen Fähigkeiten) werden unterstützt.Bibliothek
CPU
GPU
NumPy
✅
n/a
CuPy
n/a
⛔
PyTorch
✅
⛔
JAX
✅
⛔
Dask
⚠️ führt Chunks zusammen
n/a
Siehe Unterstützung für den Array API Standard für weitere Informationen.
Beispiele
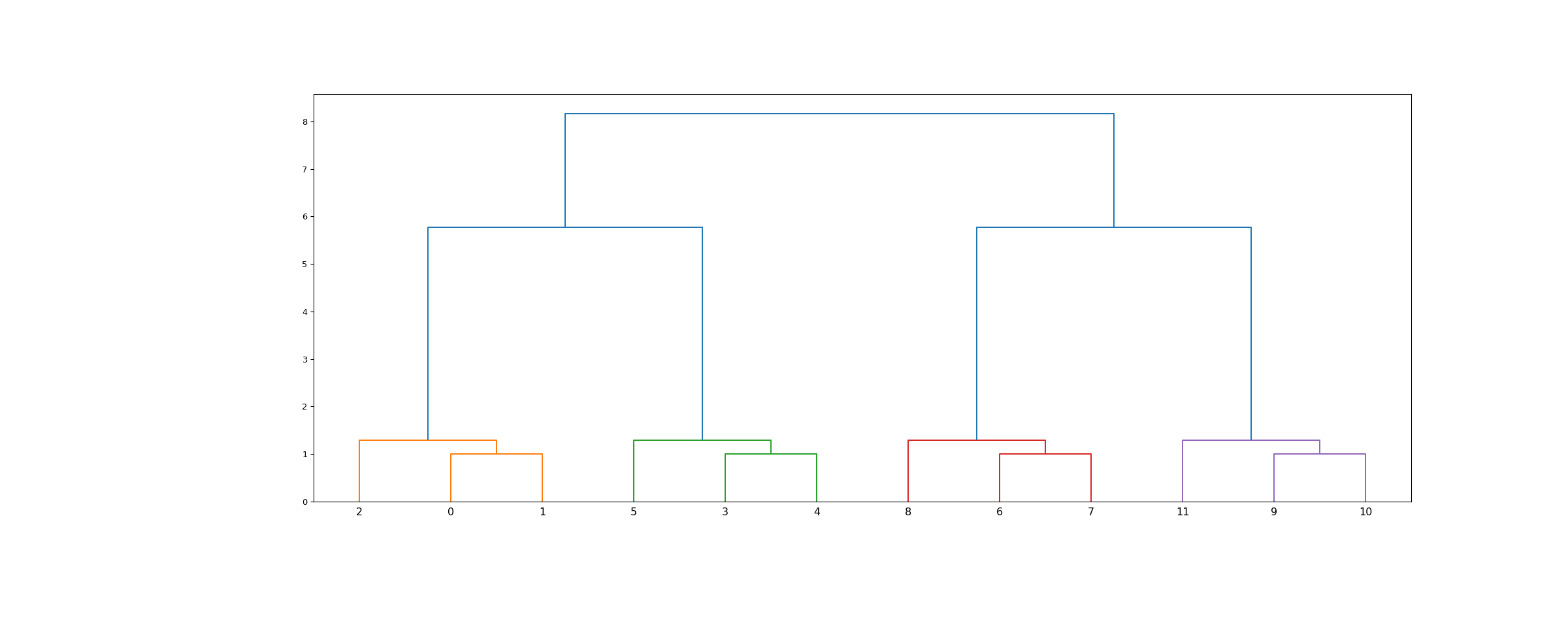
>>> from scipy.cluster.hierarchy import ward, dendrogram, leaves_list >>> from scipy.spatial.distance import pdist >>> from matplotlib import pyplot as plt
>>> X = [[0, 0], [0, 1], [1, 0], ... [0, 4], [0, 3], [1, 4], ... [4, 0], [3, 0], [4, 1], ... [4, 4], [3, 4], [4, 3]]
>>> Z = ward(pdist(X))
Die Linkage-Matrix
Zrepräsentiert ein Dendrogramm, d.h. einen Baum, der die Struktur der durchgeführten Gruppierung kodiert.scipy.cluster.hierarchy.leaves_listzeigt die Zuordnung zwischen den Indizes imXDatensatz und den Blättern im Dendrogramm>>> leaves_list(Z) array([ 2, 0, 1, 5, 3, 4, 8, 6, 7, 11, 9, 10], dtype=int32)
>>> fig = plt.figure(figsize=(25, 10)) >>> dn = dendrogram(Z) >>> plt.show()