power#
- scipy.stats.power(test, rvs, n_observations, *, significance=0.01, vectorized=None, n_resamples=10000, batch=None, kwargs=None)[Quelle]#
Simuliert die Power eines Hypothesentests unter einer alternativen Hypothese.
- Parameter:
- testcallable
Hypothesentest, dessen Power simuliert werden soll.
testmuss ein aufrufbares Objekt sein, das eine Stichprobe akzeptiert (z.B.test(sample)) oderlen(rvs)separate Stichproben (z.B.test(samples1, sample2), wenn rvs zwei aufrufbare Objekte enthält und n_observations zwei Werte enthält) und den p-Wert des Tests zurückgibt. Wenn vectorized aufTruegesetzt ist, musstestauch ein Schlüsselwortargument axis akzeptieren und vektorisiert sein, um den Test entlang der bereitgestellten axis der Stichproben durchzuführen. Jedes aufrufbare Objekt ausscipy.statsmit einem axis-Argument, das ein Objekt mit einem pvalue-Attribut zurückgibt, ist ebenfalls akzeptabel.- rvscallable or tuple of callables
Ein aufrufbares Objekt oder eine Sequenz von aufrufbaren Objekten, die Zufallsvariablen unter der alternativen Hypothese generieren. Jedes Element von rvs muss das Schlüsselwortargument
sizeakzeptieren (z.B.rvs(size=(m, n))) und ein N-dimensionales Array dieser Form zurückgeben. Wenn rvs eine Sequenz ist, muss die Anzahl der aufrufbaren Objekte in rvs mit der Anzahl der Elemente von n_observations übereinstimmen, d.h.len(rvs) == len(n_observations). Wenn rvs ein einzelnes aufrufbares Objekt ist, wird n_observations als einzelnes Element behandelt.- n_observationstuple of ints or tuple of integer arrays
Wenn eine Sequenz von Integern, jede ist die Größe einer Stichprobe, die an
testübergeben wird. Wenn eine Sequenz von Integer-Arrays, wird die Power für jeden Satz entsprechender Stichprobengrößen simuliert. Siehe Beispiele.- significancefloat or array_like of floats, default: 0.01
Die Schwelle für die Signifikanz; d.h. der p-Wert, unter dem die Ergebnisse des Hypothesentests als Beweis gegen die Nullhypothese betrachtet werden. Äquivalent dazu ist die akzeptable Rate von Typ-I-Fehlern unter der Nullhypothese. Wenn ein Array, wird die Power für jeden Signifikanzschwellenwert simuliert.
- kwargsdict, optional
Schlüsselwortargumente, die an die aufrufbaren Objekte rvs und/oder
testübergeben werden. Introspektion wird verwendet, um zu bestimmen, welche Schlüsselwortargumente an jedes aufrufbare Objekt übergeben werden können. Der Wert, der jedem Schlüsselwort zugeordnet ist, muss ein Array sein. Arrays müssen miteinander und mit jedem Array in n_observations broadcastfähig sein. Die Power wird für jeden Satz entsprechender Stichprobengrößen und Argumente simuliert. Siehe Beispiele.- vectorizedbool, optional
Wenn vectorized auf
Falsegesetzt ist, wirdtestnicht das Schlüsselwortargument axis übergeben und es wird erwartet, dass der Test nur für 1D-Stichproben durchgeführt wird. WennTrue, wirdtestdas Schlüsselwortargument axis übergeben und es wird erwartet, dass der Test entlang der axis durchgeführt wird, wenn N-dimensionale Stichproben-Arrays übergeben werden. WennNone(Standard), wird vectorized aufTruegesetzt, wennaxisein Parameter vontestist. Die Verwendung eines vektorisierten Tests reduziert typischerweise die Rechenzeit.- n_resamplesint, default: 10000
Anzahl der aus jedem der aufrufbaren Objekte von rvs gezogenen Stichproben. Äquivalent dazu ist die Anzahl der unter der alternativen Hypothese durchgeführten Tests zur Annäherung der Power.
- batchint, optional
Die Anzahl der Stichproben, die in jedem Aufruf von
testverarbeitet werden sollen. Der Speicherverbrauch ist proportional zum Produkt von batch und der größten Stichprobengröße. Standard istNone, in welchem Fall batch gleich n_resamples ist.
- Rückgabe:
- resPowerResult
Ein Objekt mit den Attributen
- powerfloat or ndarray
Die geschätzte Power gegenüber der Alternative.
- pvaluesndarray
Die unter der alternativen Hypothese beobachteten p-Werte.
Hinweise
Die Power wird wie folgt simuliert:
Zeichne viele Zufallsstichproben (oder Sätze von Stichproben), jede von der/den durch n_observations spezifizierten Größe(n), unter der durch rvs spezifizierten Alternative.
Berechne für jede Stichprobe (oder jeden Satz von Stichproben) den p-Wert gemäß
test. Diese p-Werte werden im Attributpvaluesdes Ergebnisobjekts aufgezeichnet.Berechne den Anteil der p-Werte, die kleiner als das significance-Niveau sind. Dies ist die im Attribut
powerdes Ergebnisobjekts aufgezeichnete Power.
Angenommen, significance ist ein Array der Form
shape1, die Elemente von kwargs und n_observations sind gegenseitig auf die Formshape2broadcastbar undtestgibt ein Array von p-Werten der Formshape3zurück. Dann wird das Attributpowerdes Ergebnisobjekts die Formshape1 + shape2 + shape3haben, und das Attributpvalueswird die Formshape2 + shape3 + (n_resamples,)haben.Beispiele
Angenommen, wir möchten die Power des t-Tests für unabhängige Stichproben unter den folgenden Bedingungen simulieren:
Die erste Stichprobe hat 10 Beobachtungen, die aus einer Normalverteilung mit Mittelwert 0 gezogen wurden.
Die zweite Stichprobe hat 12 Beobachtungen, die aus einer Normalverteilung mit Mittelwert 1.0 gezogen wurden.
Die Schwelle für p-Werte zur Signifikanz beträgt 0.05.
>>> import numpy as np >>> from scipy import stats >>> rng = np.random.default_rng() >>> >>> test = stats.ttest_ind >>> n_observations = (10, 12) >>> rvs1 = rng.normal >>> rvs2 = lambda size: rng.normal(loc=1, size=size) >>> rvs = (rvs1, rvs2) >>> res = stats.power(test, rvs, n_observations, significance=0.05) >>> res.power 0.6116
Mit Stichproben der Größen 10 bzw. 12 ist die Power des t-Tests bei einem Signifikanzschwellenwert von 0.05 unter der gewählten Alternative ungefähr 60%. Wir können die Auswirkung der Stichprobengröße auf die Power untersuchen, indem wir Stichprobengrößen-Arrays übergeben.
>>> import matplotlib.pyplot as plt >>> nobs_x = np.arange(5, 21) >>> nobs_y = nobs_x >>> n_observations = (nobs_x, nobs_y) >>> res = stats.power(test, rvs, n_observations, significance=0.05) >>> ax = plt.subplot() >>> ax.plot(nobs_x, res.power) >>> ax.set_xlabel('Sample Size') >>> ax.set_ylabel('Simulated Power') >>> ax.set_title('Simulated Power of `ttest_ind` with Equal Sample Sizes') >>> plt.show()
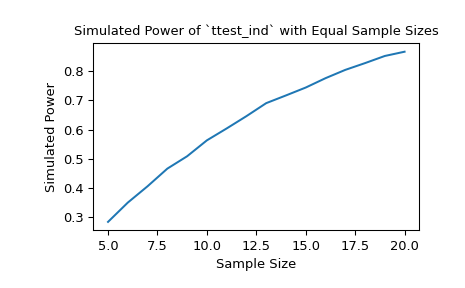
Alternativ können wir den Einfluss der Effektgröße auf die Power untersuchen. In diesem Fall ist die Effektgröße der Ort der Verteilung, die der zweiten Stichprobe zugrunde liegt.
>>> n_observations = (10, 12) >>> loc = np.linspace(0, 1, 20) >>> rvs2 = lambda size, loc: rng.normal(loc=loc, size=size) >>> rvs = (rvs1, rvs2) >>> res = stats.power(test, rvs, n_observations, significance=0.05, ... kwargs={'loc': loc}) >>> ax = plt.subplot() >>> ax.plot(loc, res.power) >>> ax.set_xlabel('Effect Size') >>> ax.set_ylabel('Simulated Power') >>> ax.set_title('Simulated Power of `ttest_ind`, Varying Effect Size') >>> plt.show()
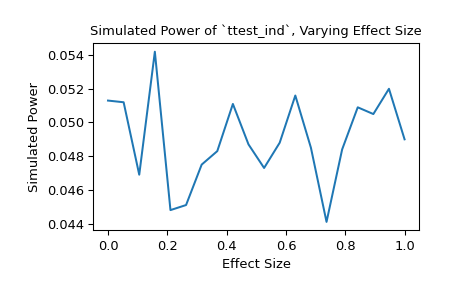
Wir können
powerauch verwenden, um die Typ-I-Fehlerrate (auch als „Größe“ bezeichnet) eines Tests zu schätzen und zu beurteilen, ob sie dem nominalen Niveau entspricht. Zum Beispiel ist die Nullhypothese vonjarque_bera, dass die Stichprobe aus einer Verteilung mit derselben Schiefe und Kurtosis wie die Normalverteilung gezogen wurde. Um die Typ-I-Fehlerrate zu schätzen, können wir die Nullhypothese als wahre *alternative* Hypothese betrachten und die Power berechnen.>>> test = stats.jarque_bera >>> n_observations = 10 >>> rvs = rng.normal >>> significance = np.linspace(0.0001, 0.1, 1000) >>> res = stats.power(test, rvs, n_observations, significance=significance) >>> size = res.power
Wie unten gezeigt, liegt die Typ-I-Fehlerrate des Tests für eine so kleine Stichprobe weit unter dem nominalen Niveau, wie in der Dokumentation erwähnt.
>>> ax = plt.subplot() >>> ax.plot(significance, size) >>> ax.plot([0, 0.1], [0, 0.1], '--') >>> ax.set_xlabel('nominal significance level') >>> ax.set_ylabel('estimated test size (Type I error rate)') >>> ax.set_title('Estimated test size vs nominal significance level') >>> ax.set_aspect('equal', 'box') >>> ax.legend(('`ttest_1samp`', 'ideal test')) >>> plt.show()
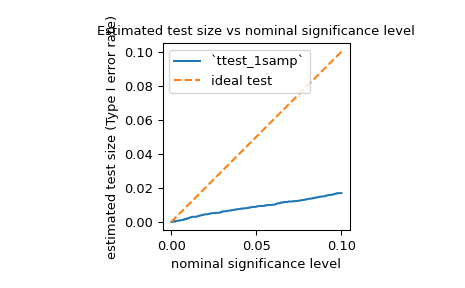
Wie man bei einem so konservativen Test erwarten kann, ist die Power gegenüber einigen Alternativen recht gering. Zum Beispiel ist die Power des Tests unter der Alternative, dass die Stichprobe aus der Laplace-Verteilung gezogen wurde, möglicherweise nicht viel größer als die Typ-I-Fehlerrate.
>>> rvs = rng.laplace >>> significance = np.linspace(0.0001, 0.1, 1000) >>> res = stats.power(test, rvs, n_observations, significance=0.05) >>> print(res.power) 0.0587
Dies ist kein Fehler in SciPy's Implementierung; es liegt einfach daran, dass die Nullverteilung der Teststatistik unter der Annahme abgeleitet wird, dass die Stichprobengröße groß ist (d.h. gegen unendlich geht), und diese asymptotische Annäherung für kleine Stichproben nicht genau ist. In solchen Fällen können Resampling- und Monte-Carlo-Methoden (z.B.
permutation_test,goodness_of_fit,monte_carlo_test) geeigneter sein.